以CRISPR-Cas9为代表的基因组编辑系统在植物研究和作物改良中已被广泛应用。然而, 常规Cas9的编辑结果主要为小片段插入/缺失 (indel), 通常无法产生多碱基的删除, 这在研究非编码RNA及调控序列如microRNA (miRNAs)、非翻译区 (UTRs) 和启动子等的功能时存在局限性。近日, JIPB在线发表了西南大学张勇课题组题为“Enhanced exonuclease-Cas9 systems enable multi-nucleotide deletions and regulatory sequence editing in rice”的研究论文 (https://doi.org/10.1111/jipb.70155)。该研究系统性开发了基于Cas9与外切核酸酶融合的多核苷酸删除Cas9 (MND-Cas9) 工具, 实现了水稻中高效多碱基删除的基因组编辑, 并展示了其在非编码RNA及调控序列功能研究中的应用潜力。
CRISPR-Cas系统是微生物进化形成的一类获得性免疫机制, 其中以CRISPR-Cas9为代表的基因组编辑技术已被发展为适用于多种生物的通用平台, 并在植物基因功能研究与作物分子育种中得到广泛应用。Cas9在向导RNA (guide RNA) 的引导下识别靶DNA序列, 并依赖PAM序列实现精确切割, 其高效性和可编程性极大推动了植物基因组定向改造技术的发展。然而, 野生型Cas9介导的编辑结果主要表现为1-2 bp的小片段插入或删除。这类突变在编码区通常足以造成基因失活, 但在非编码序列研究中往往难以产生实质性功能影响。例如, miRNA前体结构、启动子顺式调控元件以及UTR中的结构性调控模块通常需要较大范围的序列破坏才能实现有效功能干扰。因此, 如何在保持高编辑效率的同时, 获得更大尺度、更可控的多核苷酸删除突变, 成为限制CRISPR-Cas9在非编码DNA区域功能解析及精准性状改良中进一步应用的重要技术瓶颈。近年来, 研究者尝试通过与Cas9共表达或融合外切核酸酶来扩大删除范围, 但现有研究多集中于效率提升, 对编辑模式的改变缺乏系统分析, 并且其在植物非编码RNA及调控序列中的应用亦鲜有深入报道。另一方面,Cas9对NGG PAM序列的严格依赖, 也显著限制了可编辑位点的数量。尽管Cas9-NG、SpG等PAM兼容性变体的出现大幅拓宽了靶向范围, 但因其在植物中的编辑效率较低, 限制了广泛应用。
针对以上问题, 西南大学张勇教授团队通过将Trex2、SbcB等外切核酸酶及DNA结合结构域引入传统CRISPR-Cas9植物基因组编辑体系, 构建多核苷酸删除型Cas9编辑平台 (MND-Cas9), 旨在提升编辑效率的同时, 增加删除片段大小, 实现对miRNA、3’UTR等非编码DNA区域的高效破坏与功能解析, 并为复杂性状的精准分子改良提供通用化的新型基因编辑工具。实验结果表明: 1) 通过将Trex2或SbcB等外切核酸酶与Cas9融合, 成功建立第一代MND-Cas9v1系统, 在保持与Cas9相当编辑效率的前提下, 显著扩大了靶位点删除片段大小, 实现稳定的多核苷酸缺失突变; 2) 进一步引入DNA结合结构域对外切核酸酶进行空间锚定, 构建第二代MND-Cas9v2系统 (Trex2-DBD-Cas9与SbcB-DBD-Cas9), 在水稻中实现了编辑效率与删除大小的协同提升; 3) 将MND-Cas9体系与SpG及Cas9-NG等PAM宽松型变体相结合, 构建了MND-SpG和MND-Cas9-NG系统, 在显著扩展靶点覆盖范围的同时, 进一步提高了多核苷酸删除编辑的能力; 4) 以水稻miRNA基因OsmiR530及OsGhd2基因3′UTR调控元件为靶标进行功能验证, MND-Cas9v2系统高效产生了多核苷酸缺失突变, 有效破坏miRNA前体结构及SMITE的茎环结构, 显著解除其对下游基因的抑制作用。全文主要研究结果如下:
1. 植物多核苷酸删除编辑Cas9 (MND-Cas9) 的有效建立
为构建能够诱导多核苷酸删除的Cas9编辑系统
,
研究者将RecJ、T5、SbcB和Trex2四种外切核酸酶分别与SpCas9进行N端或C端融合
,
建立了8种候选MND-Cas9体系
,
并通过水稻原生质体转化
,
在6个水稻内源位点上进行了测试。结果表明
,
N端融合显著优于C端融合
,
T5-Cas9编辑效率最高 (图1B),
而SbcB-Cas9和Trex2-Cas9在保持与Cas9相当效率的同时, 显著将编辑模式由1-2 bp小缺失转变为以6-12 bp为主的多核苷酸删除, 并明显拓展了编辑范围 (图1B-D)。基于上述结果, 研究团队确立SbcB-Cas9和Trex2-Cas9为第一代多核苷酸删除编辑系统 (MND-Cas9v1)。
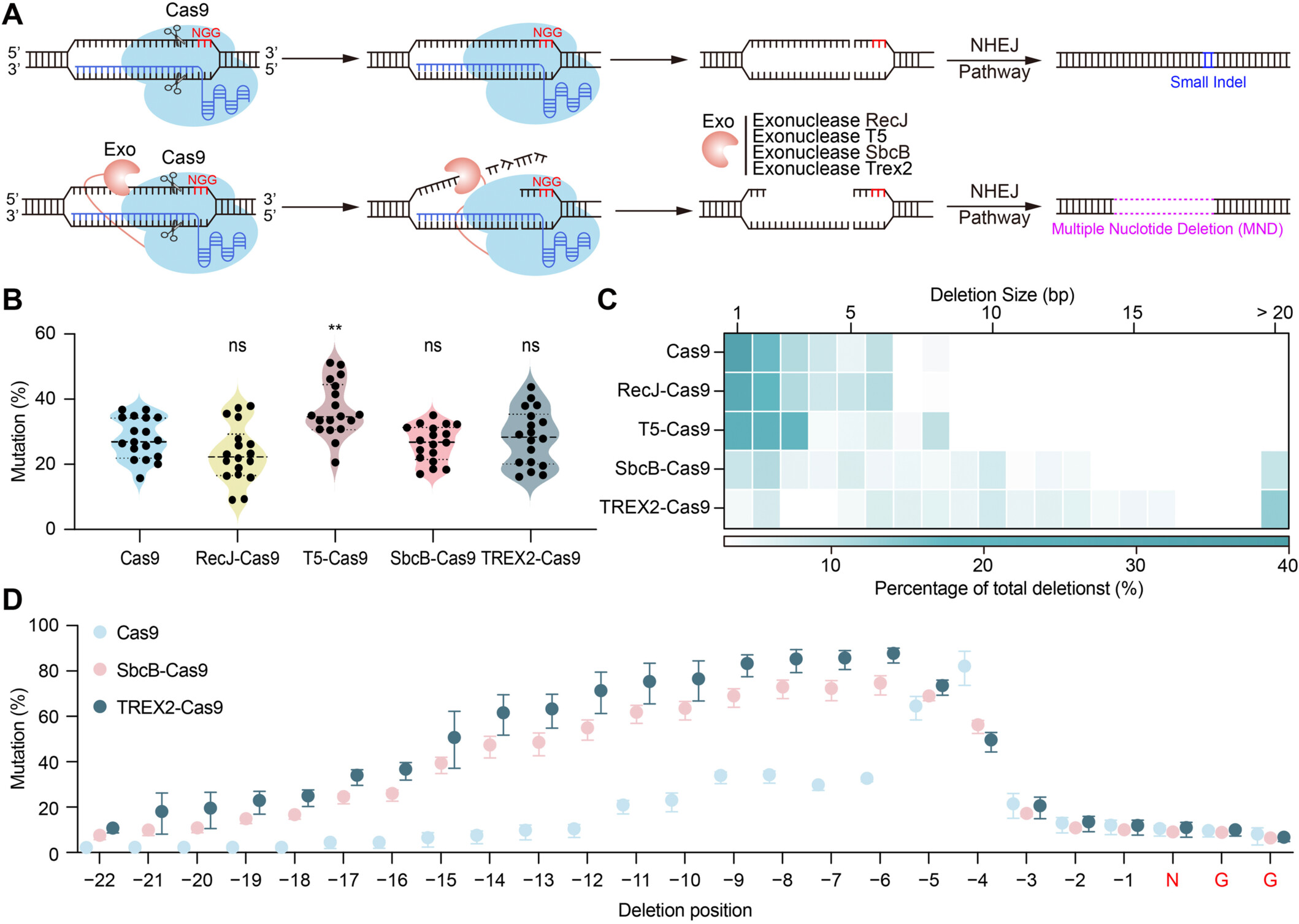
图
1 MND-Cas9系统在水稻中的编辑效率与编辑模式
2. Exo-DBD-Cas9融合建立功能增强多核苷酸缺失Cas9系统 (MND-Cas9v2)
在MND-Cas9v1系统的基础上, 研究者引入DNA结合结构域 (DBD) 将外切核酸酶锚定至Cas9附近, 以期稳定释放的非模板链, 延长外切核酸酶作用时间, 从而进一步提升编辑效率和删除大小 (图2A)。在MND-Cas9v1的基础上, 进一步构建了SbcB-DBD-Cas9和Trex2-DBD-Cas9系统 (图2B), 并在与MND-Cas9v1相同的6个水稻内源位点上进行比较。结果显示, Exo-DBD-Cas9不仅显著提升了编辑效率 (图2C), 而且显著增加了删除片段的大小: SbcB-DBD-Cas9主要产生6-14 bp缺失, Trex2-DBD-Cas9产生5-11 bp缺失 (图2D), 并将缺失位置分别扩展至PAM上游5-18 bp和5-15 bp (图2E)。总体而言, DBD融合实现了编辑效率与删除大小的协同提升, 确立了增强型多核苷酸缺失Cas9系统 (MND-Cas9v2)。
图
2 Exo-DBD-Cas9系统显著提升编辑效率与多核苷酸删除能力
3. Exo-DBD-Cas9结合PAM宽松型Cas9扩展多核苷酸删除系统可靶向范围
尽管第MND-Cas9v1/v2系统显著提升了缺失效率和范围, 但仍受限于SpCas9 NGG PAM (图3A)。为拓宽靶向范围, 研究团队将SbcB-DBD或Trex2-DBD融合至PAM宽容型Cas9变体Cas9-NG和SpG N端, 构建新的MND-Cas9v2系统 (图3B)。并通过水稻原生质体转化在8个内源位点上进行测试, 结果表明, SbcB-DBD-SpG在NGA、NGC和NCC PAM位点提升了编辑效率, Trex2-DBD-SpG在所有NGN PAM位点均稳定提升了编辑效率; Cas9-NG体系中, Trex2-DBD融合也显著提高了多种PAM位点的编辑效率 (图3C)。编辑模式分析显示, Exo-DBD-SpG和Exo-DBD-Cas9-NG显著提升了删除片段大小 (图3D), 并将缺失位置分别扩展至PAM上游5-18 bp和5-15 bp (图3E, F)。综上, PAM宽松型Exo-DBD-Cas9融合系统既克服了PAM限制, 又同步提升了编辑效率和多核苷酸删除长度, 为植物基因组编辑提供高效、广泛适用的MND工具。
图
3 PAM宽松型Exo-DBD-Cas9系统在水稻原生质体中的编辑效率与编辑模式
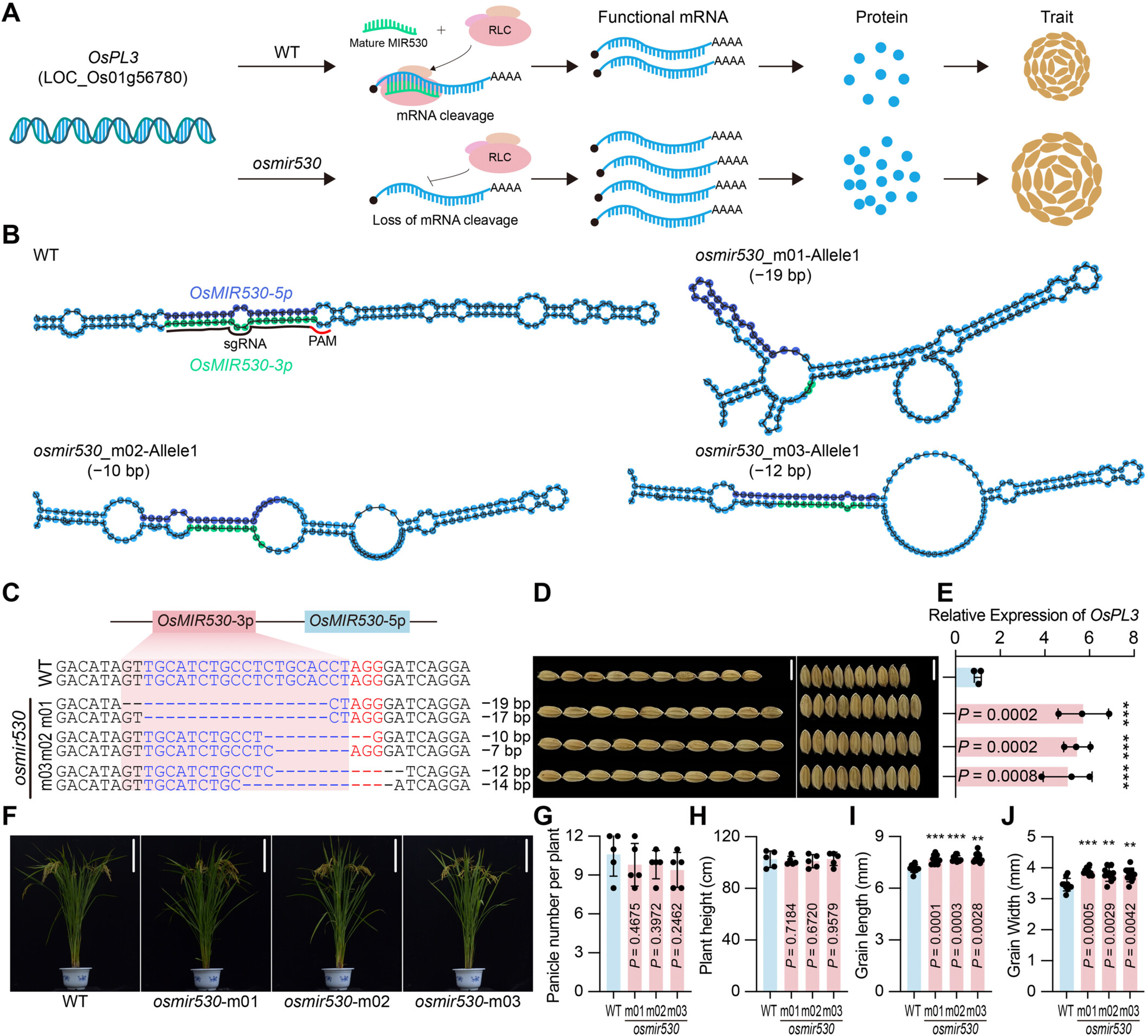
4. 基于MND-Cas9系统的OsmiR530靶向缺失及功能分析
为了进一步验证MND-Cas9在miRNA功能解析中的能力, 研究者以OsmiR530为靶标, 应用高效MND-Cas9v2系统构建编辑载体并进行水稻稳定转化 (图4A)。筛选T0植株获得三株双等位基因突变体 (osmir530-m01/02/03), 缺失长度为7-19 bp, 前体结构预测显示关键茎/环结构被破坏 (图4B, 4C), 预示成熟miRNA生物合成受阻。功能分析显示, OsmiR530失活后解除对OsPL3的负调控, 三株突变体中OsPL3表达显著上调 (图4E)。表型观察显示, 突变体株高和分蘖数与野生型无显著差异 (图4F-H), 但籽粒长度和宽度明显增加 (图4D, 4I-J)。该结果证明, MND-Cas9能够通过多核苷酸缺失高效破坏miRNA功能, 为植物miRNA调控通路解析与功能研究提供强有力工具。
图
4 MND-Cas9介导的OsmiR530靶向缺失及功能验证
5. 基于MND-Cas9系统的OsGhd2 3′UTR SMITE元件靶向编辑与功能验证
之前研究表明, OsGhd2 3′UTR中的SMITE (small miniature inverted-repeat transposable element) 序列能够通过形成稳定的茎-环结构抑制蛋白翻译 (图5A)。针对OsGhd2 3′UTR中的SMITE位点, 研究者设计了两种编辑策略: 一是利用双gRNA在SMITE上下游进行完全删除, 二是利用单gRNA靶向茎-环结构局部破坏其二级结构 (图5B), 独立编辑事件分别显示: OsGhd2-m01为SMITE全缺失, OsGhd2-m02和m03为茎-环结构部分缺失 (图5B)。表型分析显示, SMITE编辑单株株高和分蘖数与野生型无显著差异 (图5E-G), 但籽粒长度和宽度均明显增加 (图5C, H, I); qRT-PCR结果显示, OsGhd2不同类型SMITE编辑事件转录水平无显著变化 (图5D)。进一步通过双荧光报告实验验证, 在突变3′UTR背景下, ZsGreen/mCherry比值显著高于野生型3′UTR (图5J-L), 说明SMITE主要通过抑制翻译发挥作用。综合分析说明, 应用Trex2-DBD-Cas9对OsGhd2 3′UTR SMITE元件进行完整或局部删除, 解除了翻译抑制, 并改善籽粒性状, 进一步证明了MND-Cas9v2系统在解析和操控非编码DNA区域中的高效性与适用性。
图
5 MND-Cas9v2介导OsGhd2 3′UTR SMITE元件的靶向缺失及功能验证
电子科技大学生命科学与技术学院博士生张瑞、西南大学生命科学学院含弘研究员唐旭博士为论文共同第一作者, 西南大学生命科学学院/电子科技大学生命科学与技术学院张勇教授、马里兰大学植物科学与景观建筑系戚益平教授为该研究工作共同通讯作者。研究工作得到了国家科技重大专项、国家自然科学基金等项目的资助。